在正式开始介绍之前,推荐大家关注Rust for Bioinformatics这个twitter账号,最新的Rust写的生物信息学软件都会有所介绍:
如果不能够科学上网的同学们,可以关注我个人的微信平台Rust生物信息学(扫描下面的二维码),我也会更新最新的Rust生物信息学工具。
背景介绍和学习入门相关信息
计算机科学经过了一两百年的发展,从最初的底层编译语言到现在的高级语言,已经非常的成熟了,然而不可否认的是到底什么样子的高级编程语言适合什么样的工作仍然是很难回答的, 比如前端程序员,系统程序员,统计相关编程工作。比如系统级别的总是离不开C和C++,因为主流Linux的操作系统内核就是C写成的,因此不可避免使用它来进行系统层面的开发。很多流行的高级语言比如Python,R等属于脚本语言而非编译语言,速度相对而言较慢,但好处是非常方便调用, 比如计算pca 只需要一个函数,C/C++里面如果没有现成的数学计算包,还需要自己实现(实际上有很多C++数学计算库可以实现PCA)。相对而言脚本语言语法更加容易,变量定义和类别等更加概括,但牺牲的可能就是速度和更大的内存消耗。Rust作为一门非常新的语言 (2015年正式发布),吸收了几大语言的有点并且尽可能多地弥补了各自存在的缺点,这几年,尤其是2018-2022年, rust的发展速度非常快,rust团队更新的速度也非常快,几乎每六周就会有重大版本更新。
笔者2019年末开始接触Rust,被其惊人的速度所吸引,同时cargo包管理器的便捷程度也让人惊讶,相信很多人都有C/C++依赖的库安装路径的问题,究其本质,C/C++是在非互联网时代开发的语言因此库/包安装的想法仍然是基于本地的设计, 脚本语言Python和R相对则基于互联网,安装管理包都非常方便。关于Rust是如何设计的以及和C/C++相比的优点已经有很多文章介绍过,这里就不再多言。国内最好的学习Rust得资源是这个:
这本书作为入门非常合适而且讲解透彻。Rust的优点和难处在书中也解释的非常清楚,和其他语言的比较也很透彻。
本文重点探讨Rust在生物信息学和计算生物学中的应用及其优缺点。Nature 在2020年的一期技术和工具栏刊登了这样一篇文章:
讨论很多计算生物学家和生物信息学家开始转向rust的原因。最根本的其实是速度和内存/线程安全性,让多线程编程(concurrency)变得更加高效容易。内存所有权的概念是这些特性的根本。最著名的两个用于多线程编程的包是Rayon和crossbeam,第一个是把迭代直接多线程化(.iter()变成.par_iter())从而达到多线程运行的目的, crossbeam则是用于并发多线程,两者联合使用则可以快速地实现多线程编程。推荐几本笔者发现的rust多线程编程的书籍 (和上面那本入门结合使用):
安装以及CPU架构支持
安装rust通常使用rustup因为其可以方便管理不同的rust版本:The Rust toolchain installer 。只需要rustup default stable/nightly就可以在不同的rust版本之间切换。
安装rust编写的软件(只有编译后产生能运行的二进制的文件才可以被安装,否则只能作为library被使用)也非常容易,cargo install name即可,也可以直接从github page安装,比如安装graphembed: cargo install –git https://github.com/jean-pierreBoth/graphembed 即可。
不同CPU架构以及SIMD(Simple Instruction Multiple Data)支持。Rust nightly版本将intel x86_64,ARM等处理器添加了支持,因此向量化的加速非常方便。笔者的实际经验表明在intel处理器和ARM处理器上几乎无需额外的改写即可编译。
常用的生物信息学相关算法库
正如上面入门书籍提到的,从基础的语法到完成一个特定功能的软件是一段很长的路, 需要仔细研究从输入到数据的处理以及特定算法的实现,幸运的是,很多基础算法已经有算法程序员通过Rust实现了,比如k-meidoids聚类快速算法:GitHub – kno10/rust-kmedoids: k-Medoids clustering in Rust with the FasterPAM algorithm 和kmeans:https://github.com/genbattle/rkm。基础的Hash算法比如Murmurhash3也添加了rust的binding:https://github.com/stusmall/murmur3 。 最邻近搜索算法annoy的rust binding:GitHub – uzushino/rs-annoy: spotify/annoy bindings for Rust. 和rust版本的HNSW:https://gitlab.com/jpboth/hnswlib-rs 。Umap的rust版本:https://github.com/jean-pierreBoth/annembed 。跟本人研究方向相关的一些library包括 hyperloglog.rs:GitHub – tabac/hyperloglog.rs: HyperLogLog implementations. , SuperMinHash 和 probminhash:https://github.com/jean-pierreBoth/probminhash 。基因组binning算法rosella:GitHub – rhysnewell/rosella: Metagenomic Binning Algorithm 和variant calling lorikeet:GitHub – rhysnewell/Lorikeet: Strain resolver for metagenomics 。 再比如long reads correction的软件:GitHub – HudsonAlpha/rust-fmlrc 。值得一提的是,对于Single cell reads mapping和quantification,pseudoalignment用于将转录组的reads map到转录组数据库上而alevin-fry用于定量single cell sequencing的reads丰度(基于reads mapping之后的bam file, samon或者bwa等reads mapping工具)。
笔者推荐几个非常好用的rust生物信息 library, rust-bio,needletail是基础的生物序列文件读写,基础的生物信息算法比如needleman-wunsch, smith-waterman算法等还有BWT算法等都显著快于其他的语言。笔者的研究方向相关的比如genome assembler的minimzer DBG的算法实现,rust-mdbg用于PacBio长序列拼接。mathchtig用于代表kmer集合的最小文本。CoverM用于宏基因组中覆盖度的计算。再比如基于hmm预测基因组中基因的FragGeneScanRs版本达到二十倍以上的加速。最邻近搜索的kd-tree的rust实现性能也有所提升。再比如非线性降维算法t-SNE和umap也都有对应的Rust实现。所有以上提到的软件都可以在rust的库网站http://crates.io中找到。
还有一点需要提到的是,尽管rust非常高效,但很多传统的计算生物学家和生物信息学家仍然使用C/C++尤其是一些新的算法及其库,比如长序列组装的partial order alignment (POA),edit distance的库等都是C写成的,为了能够在rust中使用它们,很多喜欢用rust的同学开始重写或者写一些接口来把这些library包装起来,比如rust-spoa:GitHub – pjedge/rust-spoa: Rust crate with bindings and interface to the SPOA library for generating consensus sequences. 和edit-rs:GitHub – jean-pierreBoth/edlib-rs: rust binding to edlib 。再比如parasail序列比对的库的rust warpper parasailors: https://github.com/anp/parasailors 。
Rust并行计算
多node并行的软件包括openmpi和mpich,rsmpi是openmppi的binding,具备和openmpi一样的功能。有兴趣的同学可以仔细研究openmpi语言。
Rust和机器学习/深度学习:生物信息学中的应用
众所周知的是python的几个机器学习库接口是最方便的,然而rust也同样方便,因为tch是rust基于pytorch的接口:GitHub – LaurentMazare/tch-rs: Rust bindings for the C++ api of PyTorch.。 基于pytorch的神经网络编程也可以非常方便。基于variational autoencoder的VAMB binning 软件可以通过rust实现,笔者非常有兴趣用rust来实现VAMB软件。
Rust编辑器
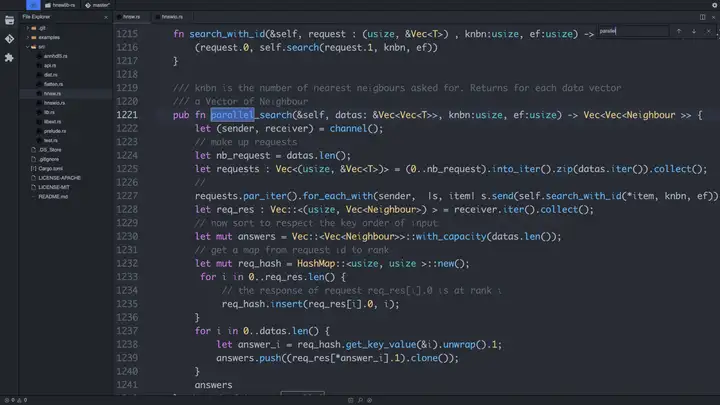
关于使用rust编程的编辑器,除了传统的Visual Studio Code等通用的编辑器,一个专门rust写成的编辑器叫做lapce:Lapce ,非常好用,速度奇快,基于rust的GPU加速,支持除了rust之外的大部分语言包括python,ruby等。由于还在开发阶段,有些功能还不具备,但是基于rust的编程工作已经非常方便快捷了。附上截图:
企业和rust
10X genomics是生物科技公司中支持rust最多的公司,10X的生物信息学家们用rust重新写很多library比如rust的DBG library rust-db和基于DBG的转录组比对pseudoalignment。大家可以关注10X genomics的github账号。
最后附上rust的图标ferris:

未完待续。
笔者暑假于亚特兰大公寓中。无聊至极欢迎骚扰。













暂无评论内容